W związku z koronawirusem COVID-19 nie dosyć, że sytuacja jest trudna, to dodatkowo chaos informacyjny w mediach nie pomaga w utrzymaniu spokoju. Dlatego czas wziąć sprawę w swoje ręce i za pomocą pakietu MATLAB przyglądnąć się bliżej wiarygodnym danym dotyczącym rozprzestrzeniania się choroby.
MATLAB, COVID-19 i Uniwersytet John’a Hopkins’a
Pod koniec stycznia 2020 roku, Uniwersytet John’a Hopkins’a w Baltimore rozpoczął agregację danych dotyczących konsekwencji rozprzestrzeniania się koronawirusa COVID-19 na Świecie. W repozytorium znajdującym się na portalu github.com są codziennie zapisywane dane dotyczące liczby osób u których wykryto wirusa, liczby zgonów i liczby wyzdrowiałych. Dane pochodzą od różnych organizacji (ich lista znajduje się na stronie repozytorium) i dotyczą niemal wszystkich państw, w których prowadzi się tego typu statystyki. Ta baza danych jest dostępna publicznie i można z niej korzystać w celach niekomercyjnych.
Zadanie postawione przed MATLAB 'em jest proste: pobierz bazę danych Uniwersytetu John’a Hopkins’a z Internetu, zapisz na dysku, przekonwertuj do postaci tabeli i wykonaj prostą analizę danych, porównując statystyki z Polski i dwóch innych wybranych krajów Europy. Następnie narysuj rysunki obrazujące trendy w liczbie osób zakażonych, zgonów i liczby osób wyzdrowiałych. Oblicz procentową śmiertelność w danym kraju. Zaczynamy!
Program poniżej powstał na bazie skryptu E. Cheynet'a, który jest dostępny w ‘MATLAB Exchange’. Parametry importu danych zostały ustawione z wykorzystaniem obiektu delimitedTextImportOptions. Jest to względnie nowa możliwość w MATLABie, obecna od 2016 roku i wersji programu R2016b. Ścieżka do repozytorium jest przechowywana w zmiennej address, a odczyt danych następuje za pomocą funkcji urlwrite. Następnie są tworzone trzy tabele zawierające dane dotyczące osób zakażonych, zgonów i osób wyleczonych. Analizę danych można prowadzić na różne sposoby w zależności od tego jakie informacje czy charakterystyki nas interesują. W tym wpisie ograniczę się do najprostszych wykresów obrazujących trendy w liczbie danych przypadków, ich udziału procentowego w całej populacji oraz obliczę śmiertelność choroby wywoływanej przez koronawirus COVID-19.
Program znajdziecie na końcu wpisu. Teraz zobaczmy jakie rysunki możemy uzyskać i co z nich wynika. Ponieważ w skali liniowej trudno zobaczyć niewielkie zmiany trendu, więc większość rysunków jest w skali logarytmicznej. Ma to ten walor, że dobrze obrazuje przyrost wykładniczy - jeżeli z takim przyrostem mamy do czynienia to linia wykresu jest prosta.
Polska
Poniższe rysunki obrazują trend przyrostów osób zakażonych, wyleczonych i zgonów. Widać, że w skali logarytmicznej dostajemy czytelniejszy rysunek.
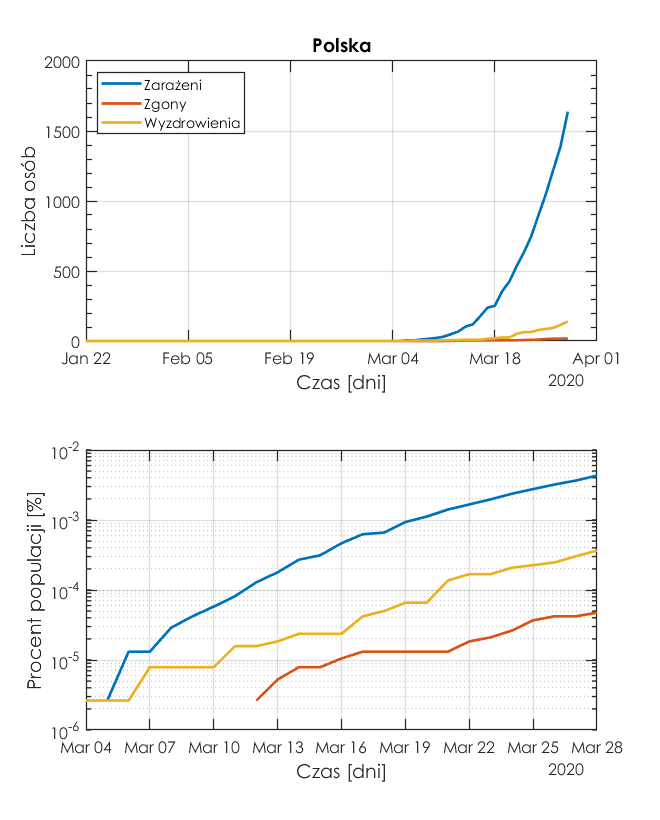
Jak widać trend wszystkich wskaźników jest wciąż wzrostowy. Porównajmy liczbę zarażonych w Polsce z przypadkami we Włoszech i Szwajcarii.
Liczba zarażonych
Poniżej rysunki w skali logarytmicznej. Górny przedstawia liczbę zakażonych osób, a dolny stosunek liczby zakażonych do liczności populacji w danym kraju - jest to więc procent osób zainfekowanych.
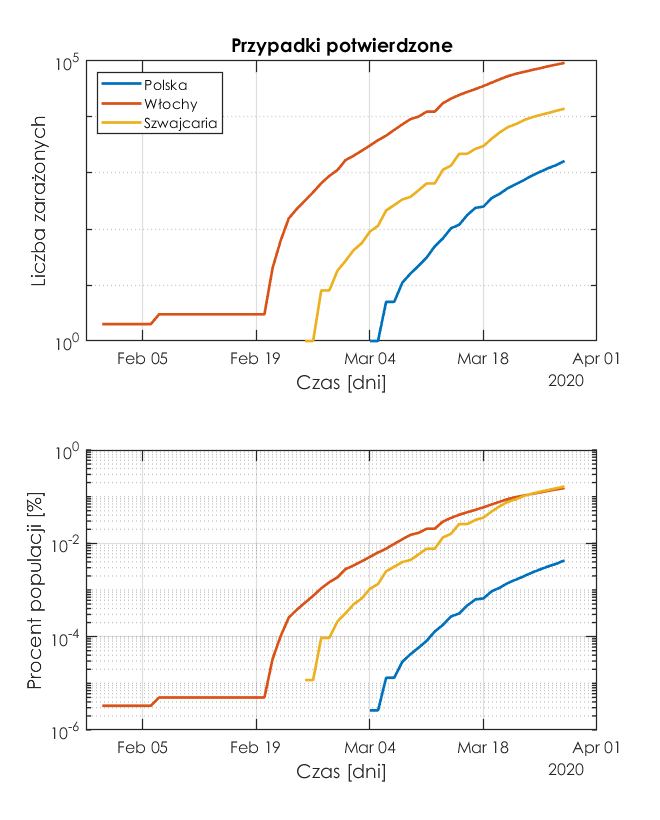
Z rysunku wynikają trzy zasadnicze wnioski:
- W różnych krajach przyrost liczby zakażeń zaczął się w różnych okresach.
- Największy odsetek zarażonych względem całej populacji kraju jest we Włoszech i Szwajcarii, a najmniejszy w Polsce.
- Trudno na podstawie wykresu wnioskować na temat tempa wzrostu (liczby zakażeń na dzień).
Tempo wzrostu zakażeń na dzień
Aby sprawdzić, w którym kraju tempo zachorowań jest największe, należy policzyć pochodną z liczby stwierdzonych przypadków. MATLAB ma do tego funkcję diff. W ten sposób dostajemy tempo zakażeń, czyli liczbę wykrytych przypadków na jeden dzień. Oczywiście wpływ na ten obraz ma liczba przeprowadzanych testów, ale ta zmienna jest poza naszą wiedzą.
Poniżej są rysunki w skali liniowej i logarytmicznej.
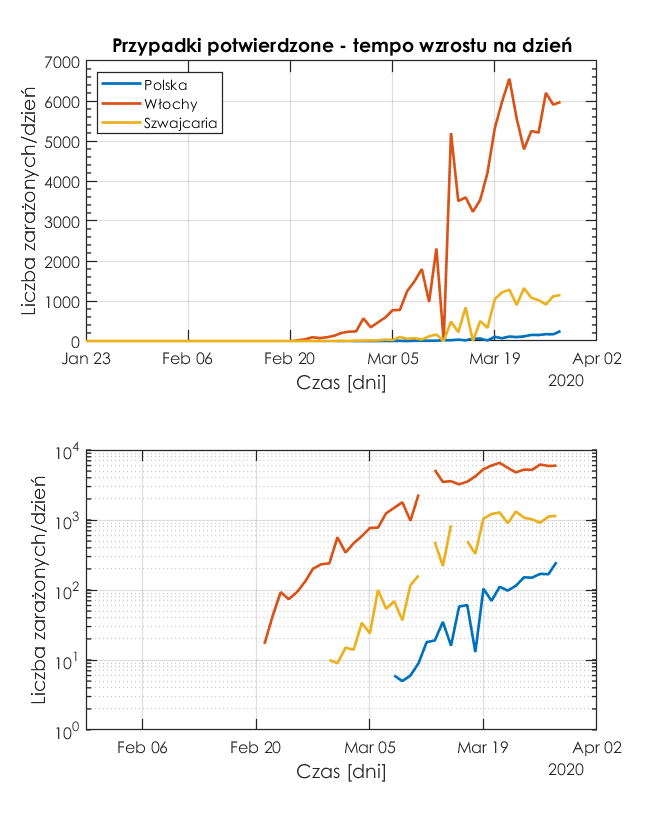
Z rysunku wynika, że największe tempo zakażeń występuje we Włoszech, a najmniejsze w Polsce.
Zgony
Poniżej są rysunki w skali logarytmicznej. Górny przedstawia liczbę zgonów, a dolny stosunek liczby zgonów do liczności populacji w danym kraju - jest to więc procent populacji, która zmarła w z powodu koronawirusa. .
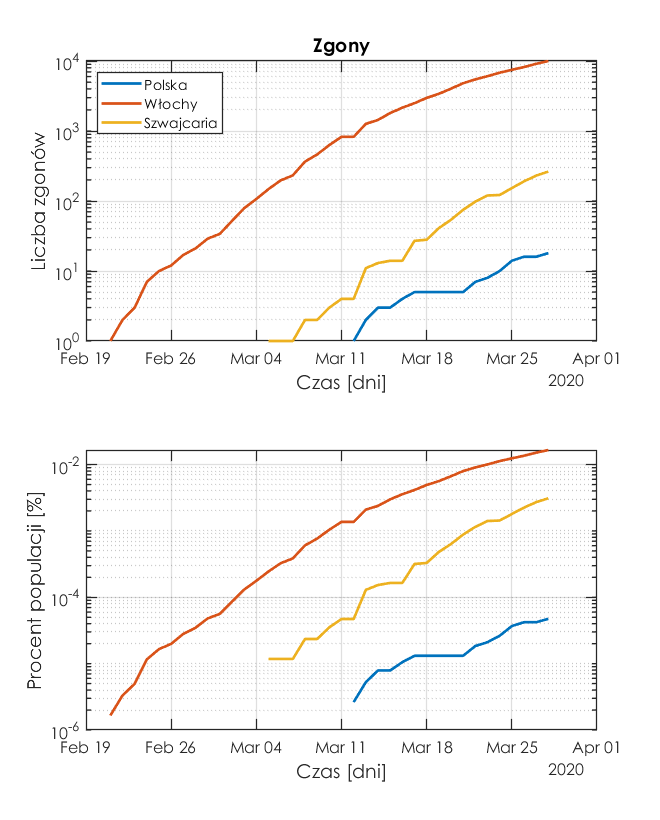
Policzmy tempo wzrostu zgonów. Ta charakterystyka wydaje się być bardziej wiarygodna od tempa wzrostu zakażeń, gdyż nie ma tu zmiennej w postaci liczby i efektywności testów wykrywających koronawirus COVID-19.
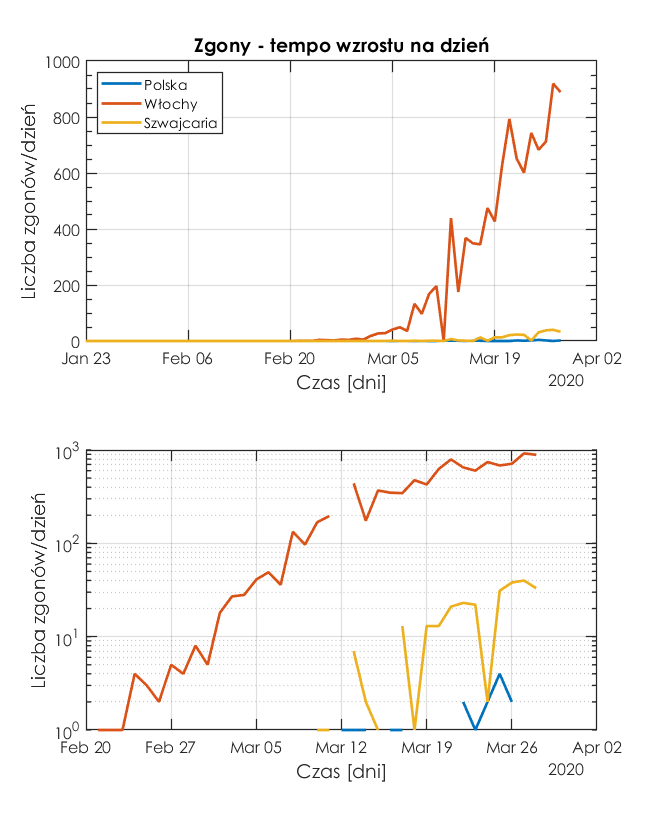
Z rysunku wynika, że największy dzienny przyrost zgonów jest we Włoszech, a najmniejszy w Polsce. Ponieważ w skali logarytmicznej nie da się zobrazować wartości '0', to w wykresach "brakuje" danych, jeżeli przyrost wynosił 0.
Śmiertelność
Te rozważania prowadzą nas do kolejnej smutnej charakterystyki.
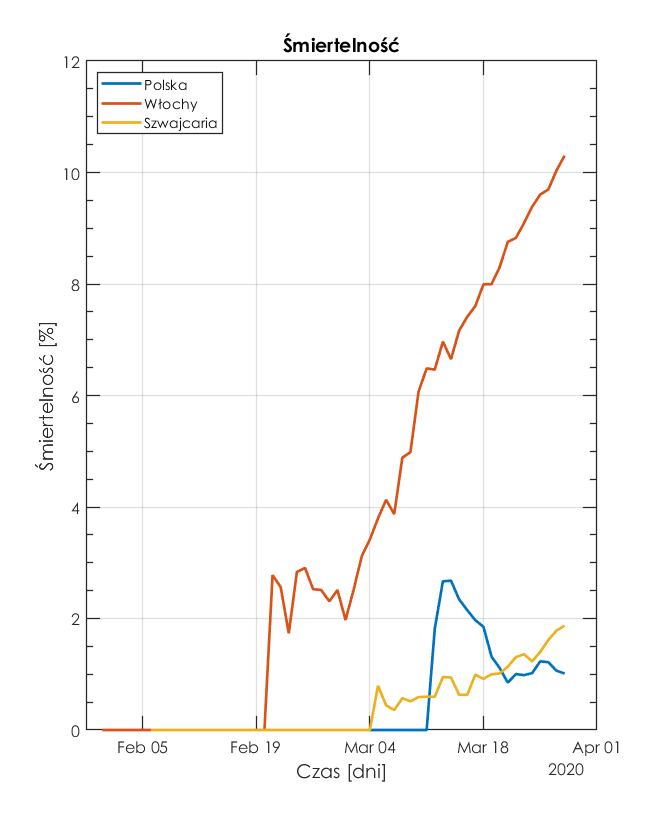
Obecna śmiertelność we Włoszech wynosi 10%. W Polsce i Szwajcarii, śmiertelność jest na poziomie 1%.
Wyzdrowienia
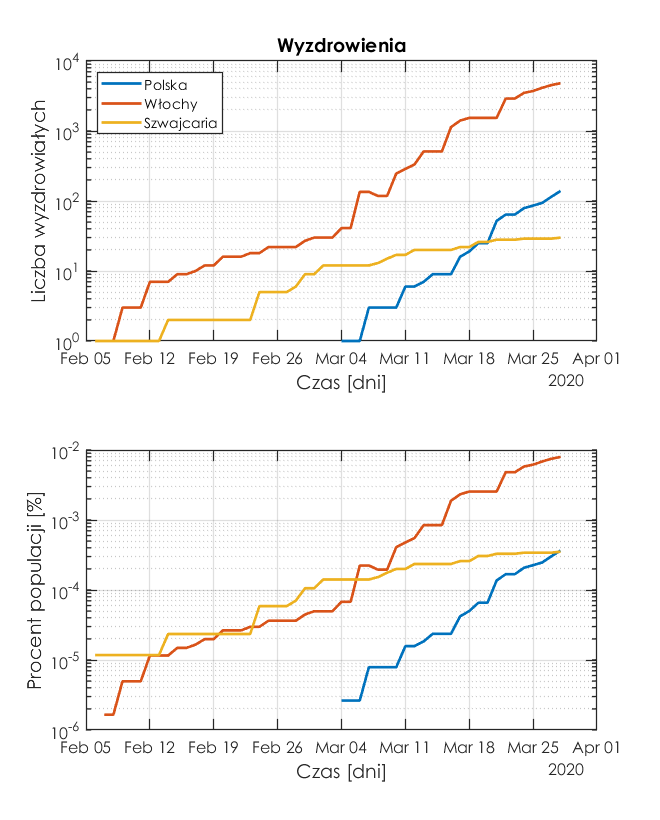
Jest to najbardziej optymistyczna charakterystyka, choć w Szwajcarii tempo wyzdrowień utrzymuje się na stałym poziomie i pod tym względem "doganiamy" Szwajcarów.
Podsumowanie
Już prosta analiza danych daje ciekawy obraz rozwoju pandemii koronawirus COVID-19. MATLAB dzięki możliwości wczytywania danych bezpośrednio z repozytorium John’a Hopkins’a, daje nam możliwość wygodnej analizy sytuacji i monitorowania rozwoju choroby na Świecie.
W kolejnym wpisie zajmę się bardziej wyszukaną analizą danych związanych z koronawirusem i spróbuję odpowiedzieć na pytanie, jakie potencjalne scenariusze są przed nami i "ile to jeszcze potrwa"?
Program
clear all; close all; clc
% Program na podstawie skryptu E. Cheynet'a i funkcji:
% [tableConfirmed,tableDeaths,tableRecovered,time] = getDataCOVID()
% Program pobiera dane dotyczące liczby zachorowań na COVID-19 z bazy
% danych uniwersytetu John'a Hopkins'a oraz wykonuje prostą analizę danych.
%
% Referencje:
% [1] https://github.com/CSSEGISandData/COVID-19
%% Parametry importu danych
% Liczba dni do analizy od początku rejestracji danych
Ndays = floor(datenum(now))-datenum(2020,01,22)-1; % minus jeden dzień bo baza danych jest odświeżana co 24h
% Ustawienie parametrów importu danych
opts = delimitedTextImportOptions("NumVariables", Ndays+5);
% Dzięki użyciu tabeli, każdej zmiennej można nadać nazwę symboliczną
% i się nią posługiwać w programie
opts.VariableNames = ["ProvinceState", "CountryRegion", "Lat", "Long", repmat("data",1,Ndays+1)];
% Definicja typu każdej zmiennej, dwie pierwsze to stringi, reszta wartości numeryczne:
opts.VariableTypes = ["string", "string", repmat("double",1,Ndays+3)];
% Pozostałe ustawienia importu danych
opts.ExtraColumnsRule = "ignore"; % w przypadku występowania dodatkowych kolumn - ignorujemy je
opts.EmptyLineRule = "read"; % jeżeli w danych występują puste linie to je odczytujemy
%% Import danych
% Dane podzielone na trzy grupy: zarażeni, zgony i wyleczenia
status = {'confirmed','deaths','recovered'};
% Adres repozytorium Uniwersytetu John'a Hopkins'a
address = 'https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/';
ext = '.csv';
% Odczyt danych
for ii = 1:numel(status)
filename = ['time_series_covid19_',status{ii},'_global'];
fullName = [address,filename,ext];
urlwrite(fullName,'dummy.csv'); % import danych do pliku, którego zawartość
% będzie następnie konwertowana na tabelę:
if strcmpi(status{ii},'Confirmed')
tableConfirmed =readtable('dummy.csv', opts);
elseif strcmpi(status{ii},'Deaths')
tableDeaths =readtable('dummy.csv', opts);
elseif strcmpi(status{ii},'Recovered')
tableRecovered =readtable('dummy.csv', opts);
else
error('Unknown status')
end
end
% Odczyt daty i jej konwersja na postać standardową
fid = fopen('dummy.csv');
time = textscan(fid,repmat('%s',1,size(tableRecovered,2)), 1, 'Delimiter',',');
time(1:4)=[];
time = datetime([time{1:end}])+years(2000);
fclose(fid);
% delete('dummy.csv'); % plik można skasować lub zostawić (przydatne przy
% braku połączenia z Internetem
%% Analiza danych
% Dane dla Polski
Poland_population = 38*10^6;
Poland_confirmed = tableConfirmed(tableConfirmed.CountryRegion == 'Poland',:);
Poland_deaths = tableDeaths(tableConfirmed.CountryRegion == 'Poland',:);
Poland_recovered = tableRecovered(tableConfirmed.CountryRegion == 'Poland',:);
% Procent zachorowań w stosunku do liczby mieszkańców
Poland_conf_ratio = 100*Poland_confirmed{:,5:end}/Poland_population;
Poland_deaths_ratio = 100*Poland_deaths{:,5:end}/Poland_population;
Poland_recovered_ratio = 100*Poland_recovered{:,5:end}/Poland_population;
% Śmiertelność
Poland_mortality_ratio = 100*Poland_deaths{:,5:end}./(Poland_recovered{:,5:end}+Poland_confirmed{:,5:end});
% Dane dla kraju nr 1
Country1 = 'Italy'
Country1_population = 60.4*10^6;
Country1_confirmed = tableConfirmed(tableConfirmed.CountryRegion == Country1,:);
Country1_deaths = tableDeaths(tableConfirmed.CountryRegion == Country1,:);
Country1_recovered = tableRecovered(tableConfirmed.CountryRegion == Country1,:);
% Procent zachorowań w stosunku do liczby mieszkańców
Country1_conf_ratio = 100*Country1_confirmed{:,5:end} / Country1_population;
Country1_deaths_ratio = 100*Country1_deaths{:,5:end} / Country1_population;
Country1_recovered_ratio = 100*Country1_recovered{:,5:end} / Country1_population;
% Śmiertelność
Country1_mortality_ratio = 100*Country1_deaths{:,5:end}./(Country1_recovered{:,5:end}+Country1_confirmed{:,5:end});
% Dane dla kraju nr 2
Country2 = 'Switzerland'
Country2_population = 8.5*10^6;
Country2_confirmed = tableConfirmed(tableConfirmed.CountryRegion == Country2,:);
Country2_deaths = tableDeaths(tableConfirmed.CountryRegion == Country2,:);
Country2_recovered = tableRecovered(tableConfirmed.CountryRegion == Country2,:);
% Procent zachorowań w stosunku do liczby mieszkańców
Country2_conf_ratio = 100*Country2_confirmed{:,5:end} / Country2_population;
Country2_deaths_ratio = 100*Country2_deaths{:,5:end} / Country2_population;
Country2_recovered_ratio = 100*Country2_recovered{:,5:end} / Country2_population;
% Śmiertelność
Country2_mortality_ratio = 100*Country2_deaths{:,5:end}./(Country2_recovered{:,5:end}+Country2_confirmed{:,5:end});
% Tempo przyrostu zakażeń
delta_Poland = diff(Poland_confirmed{1,5:end});
delta_Country1 = diff(Country1_confirmed{1,5:end});
delta_Country2 = diff(Country2_confirmed{1,5:end});
% Tempo przyrostu zgonów
delta_deaths_Poland = diff(Poland_deaths{1,5:end});
delta_deaths_Country1 = diff(Country1_deaths{1,5:end});
delta_deaths_Country2 = diff(Country2_deaths{1,5:end});
%% Rysunki
% Polska w skali liniowej
figure; subplot(211); plot(time, Poland_confirmed{:,5:end}, time, Poland_deaths{:,5:end}, time, Poland_recovered{:,5:end})
my_figure('Czas [dni]', 'Liczba osób', 'Polska'); grid on;
legend('Zarażeni','Zgony','Wyzdrowienia','Location','northwest')
subplot(212); semilogy(time, Poland_conf_ratio, time, Poland_deaths_ratio, time, Poland_recovered_ratio)
my_figure('Czas [dni]', 'Procent populacji [%]', ' '); grid on;
% Polska w skali logarytmicznej
figure; subplot(211); semilogy(time, Poland_confirmed{:,5:end}, time, Poland_deaths{:,5:end}, time, Poland_recovered{:,5:end})
my_figure('Czas [dni]', 'Liczba osób', 'Polska'); grid on;
legend('Zarażeni','Zgony','Wyzdrowienia','Location','northwest')
subplot(212); semilogy(time, Poland_conf_ratio, time, Poland_deaths_ratio, time, Poland_recovered_ratio)
my_figure('Czas [dni]', 'Procent populacji [%]', ' '); grid on;
% Zarażeni w skali liniowej
figure; subplot(211); plot(time, Poland_confirmed{:,5:end}, time, Country1_confirmed{:,5:end}, time, Country2_confirmed{:,5:end})
my_figure('Czas [dni]', 'Liczba zarażonych', 'Przypadki potwierdzone'); grid on;
legend('Polska','Włochy','Szwajcaria','Location','northwest')
subplot(212); plot(time, Poland_conf_ratio, time, Country1_conf_ratio, time, Country2_conf_ratio)
my_figure('Czas [dni]', 'Procent populacji [%]', ' '); grid on;
% Zarażeni w skali logarytmicznej
figure; subplot(211); semilogy(time, Poland_confirmed{:,5:end}, time, Country1_confirmed{:,5:end}, time, Country2_confirmed{:,5:end})
my_figure('Czas [dni]', 'Liczba zarażonych', 'Przypadki potwierdzone'); grid on;
legend('Polska','Włochy','Szwajcaria','Location','northwest')
subplot(212); semilogy(time, Poland_conf_ratio, time, Country1_conf_ratio, time, Country2_conf_ratio)
my_figure('Czas [dni]', 'Procent populacji [%]', ' '); grid on;
% Szybkość zakażeń
figure; subplot(211); plot(time(2:end), delta_Poland, time(2:end), delta_Country1, time(2:end), delta_Country2)
my_figure('Czas [dni]', 'Liczba zarażonych/dzień', 'Przypadki potwierdzone - tempo wzrostu na dzień'); grid on;
legend('Polska','Włochy','Szwajcaria','Location','northwest')
subplot(212); semilogy(time(2:end), delta_Poland, time(2:end), delta_Country1, time(2:end), delta_Country2)
my_figure('Czas [dni]', 'Liczba zarażonych/dzień', ' '); grid on;
% Zgony w skali logarytmicznej
figure; subplot(211); semilogy(time, Poland_deaths{:,5:end}, time, Country1_deaths{:,5:end}, time, Country2_deaths{:,5:end})
my_figure('Czas [dni]', 'Liczba zgonów', 'Zgony'); grid on;
legend('Polska','Włochy','Szwajcaria','Location','northwest')
subplot(212); semilogy(time, Poland_deaths_ratio, time, Country1_deaths_ratio, time, Country2_deaths_ratio)
my_figure('Czas [dni]', 'Procent populacji [%]', ' '); grid on;
% Szybkość zgonów
figure; subplot(211); plot(time(2:end), delta_deaths_Poland, time(2:end), delta_deaths_Country1, time(2:end), delta_deaths_Country2)
my_figure('Czas [dni]', 'Liczba zgonów/dzień', 'Zgony - tempo wzrostu na dzień'); grid on;
legend('Polska','Włochy','Szwajcaria','Location','northwest')
subplot(212); semilogy(time(2:end), delta_deaths_Poland, time(2:end), delta_deaths_Country1, time(2:end), delta_deaths_Country2)
my_figure('Czas [dni]', 'Liczba zgonów/dzień', ' '); grid on;
% Śmiertelność w skali logarytmicznej
figure; plot(time, Poland_mortality_ratio, time, Country1_mortality_ratio, time, Country2_mortality_ratio)
my_figure('Czas [dni]', 'Śmiertelność [%]', 'Śmiertelność'); grid on;
legend('Polska','Włochy','Szwajcaria','Location','northwest')
% Wyzdrowieni w skali logarytmicznej
figure; subplot(211); semilogy(time, Poland_recovered{:,5:end}, time, Country1_recovered{:,5:end}, time, Country2_recovered{:,5:end})
my_figure('Czas [dni]', 'Liczba wyzdrowiałych', 'Wyzdrowienia'); grid on;
legend('Polska','Włochy','Szwajcaria','Location','northwest')
subplot(212); semilogy(time, Poland_recovered_ratio, time, Country1_recovered_ratio, time, Country2_recovered_ratio)
my_figure('Czas [dni]', 'Procent populacji [%]', ' '); grid on;
function my_figure(opisX, opisY, tytul)
%% Zmienione argumenty wejściowe funkcji na opisy osi!
%% CREATEFIGURE2(X1, Y1) <-- Taka była pierwotna składnia funkcji.
% X1: vector of x data
% Y1: vector of y data
% Auto-generated by MATLAB on 18-Mar-2020 10:38:53
% Create figure
% figure1 = figure; % To usuwamy, gdyż rysunek będzie tworzony
% na zewnątrz funkcji
% Create axes
% axes1 = axes; % To również usuwamy, gdyż obiekt axes, jest już
% stworzony na aktywnym rysunku
axes1 = gca;
hold(axes1,'on');
r = groot;
monitors = r.MonitorPositions;
figure1 = gcf;
figure1.Position = [monitors(1,1)+200 monitors(2,4)/5 660 820];
% Create plot
% plot(X1,Y1,'LineWidth',2); % To samo dotyczy linii
% Create ylabel
% ylabel({'Opis osi Y'}); % Podmieniamy, tak aby móc wprowadzać
ylabel(opisY); % swój tekst. Podobnie z xlabel i title
% Create xlabel
xlabel(opisX);
% Create title
title(tytul);
% Uncomment the following line to preserve the X-limits of the axes
% xlim(axes1,[0 1]);
box(axes1,'on');
% Set the remaining axes properties
set(axes1,'FontName','Century Gothic','FontSize',12,...
'LabelFontSizeMultiplier',1.2,'TitleFontSizeMultiplier',1.2,'XGrid','on',...
'LineWidth',1,'TickLength',[0.02 0.025],'XMinorTick','on','YGrid','on','YMinorTick','on');
%% Jeżeli rysunek zawiera więcej niż jedną linia, konieczna jest pętla:
liczba_lini = size(axes1.Children);
for k = 1 : liczba_lini
axes1.Children(k,1).LineWidth = 2;
end
end

Super, dzięki.
Ale jest problem w linii:
%figure1.Position = [monitors(1,1)+200 monitors(2,4)/5 660 820];
Kwestia liczby monitorów i ich rozdzielczości. Ta linia ustawia rysunek w miejscu, w którym chce na moich monitorach. Jeżeli jest z tym problem, proszę to usunąć, a rysunki pojawią się w domyślnym miejscu.
Tak właśnie zrobiłem, dzięki i czekam na cześć II
Hej!
Jak rozwiązać problem?
Error using urlreadwrite (line 98)
Error downloading URL. Your network connection may be down or your proxy settings improperly
configured.
Error in urlwrite (line 52)
[f,status] = urlreadwrite(mfilename,catchErrors,url,filename,varargin{:});
Error in covid19 (line 18)
urlwrite(fullName,'dummy.csv');
'Your network connection may be down or your proxy settings improperly configured' czyli problem leży po stronie połączenia z Internetem albo serwer jest chwilowo przeciążony. Od czasu do czasu to się zdarza i nie ma nic wspólnego z programem.